- TOP
- >
- 膵癌の早期発見に希望 膵癌バイオマーカー開発
新着ニュース30件
2016年6月8日 20:00
血中の反復配列RNAの高感度測定法
2016年6月3日、東京大学医学部附属病院・岸川孝弘特任臨床医、大塚基之助教、小池和彦教授の研究グループは、これまで測定困難だった、膵癌患者および前癌病態患者の血液中に高頻度に検出される、反復配列RNAといわれる特殊なRNA定量法の開発に成功したことを発表した。背景
現在日本において、膵癌は癌死因の第4位になっている。膵癌は早期診断が難しく、外科的切除ができない高度進行状態で見つかる場合が多いのが現状である。高感度かつ検診レベルで導入可能な低侵襲・低コストなバイオマーカーの開発が急がれている。膵癌およびその前癌病態でHSATIIとよばれるゲノム領域から転写される反復配列RNAが異常高発現していることが報告されている。HSATIIを血中で検出することができれば、有力なバイオマーカーとなると考えられるものの、検診レベルで導入できる簡便かつ正確な定量は困難だった。
研究成果
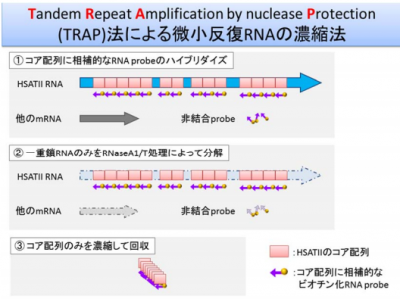
今回の研究において、HSATII RNAを高感度に定量するための方法として、TRAP法(Tandem Repeat Amplification法)を開発した。TRAP法は、反復配列RNAのコアとなる配列に相補的なprobeをハイブリダイズさせた後に、二重鎖以外のRNAを分解し、コア配列のみを濃縮し回収する方法である。その後、サンプルを、微量検体を正確に増幅して定量できるdroplet digital PCR法を用いて定量することで、RNA を高感度に捉えることができる。
研究グループは実際の膵癌患者の血清400μlからのHSATIIRNAの検出について検討した結果、血中HSATII RNAの測定は膵癌患者の早期診断を目的としたスクリーニングだけでなく、膵癌の前癌病態の囲い込みにも有用である可能性が示唆された。今後、HSATII RNA検出法が普及し、膵癌の早期診断が可能になることが期待される。
(画像はプレスリリースより)
東京大学 プレスリリース
http://www.h.u-tokyo.ac.jp/vcms_lf/release_20160603-2.pdf
-->